DNA na dnie
W czwartek, korzystając ze zorganizowanej wycieczki szkolnej – zostałem zaproszony na zwiedzenie w Centrum Onkologii, gliwickiego oddziału – Instytutu im. Marii Skłodowskiej-Curie a dokładnie zakładu biologii nowotworów.
Tam Kasia Lisowska pokazała jak można w próbówce zawierającej DNA myszy [wyizolowanego z ogona] sprawić, by samo DNA było widoczne.
Dodać trzeba było 10 μl tego i 300 μl tamtego, zamieszać chwilę i już… DNA myszy jest widoczne na dnie tej próbówki jako biała pogięta nitka:

Potem bawiliśmy się w żelu.
Najpierw mieszaliśmy — powielone (amplifikowane) wcześniej metodą łańcuchowej reakcji polimerazy (PCR – polymerase chain reaction) –fragmenty DNA z barwnikiem, które daje się na płytki żelowe.
Płytka żelowa umieszczona była w małym pojemniku do którego podłączony był transformator.
DNA ma ładunek ujemny i zgodnie z tym ładunkiem, migruje przez żel w kierunku anody. Dzięki barwnikowi mniej więcej widać dokąd DNA ,,zawędrowało”.
Nazywa się to elektroforeza i dobrze jest opisane tu.
Żeby uwidocznić DNA, do żelu dodany jest barwnik (np. bromek etydyny), który wiąże się z DNA. Bromek etydyny świeci na pomarańczowo w świetle ultrafioletowym – dzięki temu można zobaczyć DNA.
Bromek etydyny jest mutagenny i rakotwórczy, więc żele z bromkiem trzeba przenosić w rękawiczkach ochronnych.
Następnie taki żel został umieszczony w komorze z promieniowaniem UV, które uwidoczniło kawałki DNA. Tam też zrobione było zdjęcie, które po wydrukowaniu może wyglądać tak:
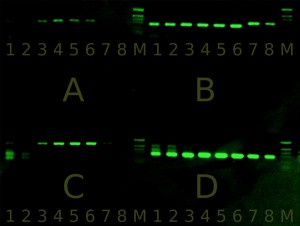
Na zdjęciu żelu widać produkty czterech różnych reakcji, każda z reakcji była przeprowadzana w rosnącej temperaturze przyłączania starterów. Dzięki temu można wyznaczyć odpowiednią temperaturę, czyli na tyle wysoką, że nie powstają w niej niespecyficzne produkty (widoczne jako dodatkowe prążki na żelu) i na tyle niską, że wciąż jeszcze powstaje pożądany produkt. Najlepiej to widać w części D.
M oznacza marker wielkości, czyli mieszaninę fragmentów DNA o znanej wielkości. Przez porównanie można ocenić wielkość produktu reakcji PCR.
W ścieżkach od 1 do 8 mamy produkty reakcji PCR wykonywanej w temperaturze przyłączania starterów od 50°C do 68°C (co 2 stopnie).
W temperaturach 50-54°C (ścieżki 1, 2, 3) oprócz właściwego produktu powstały też większe od niego produkty niespecyficzne. W Temperaturach od 56-64°C został wydajnie syntezowany właściwy produkt, a w temp. 66 i 68°C zaczyna spadać wydajność reakcji (prążek jest słabszy), bo temperatura wiązania starterów do matrycy jest już zbyt wysoka.
Z tego eksperymentu wynika, że dla wydajnej amplifikacji interesującego nas fragmentu DNA najlepszy jest zakres temperatur od 56°C — do 64°C.
I po tej wizycie mam obrazek niczym na biurko.




Dodaj komentarz